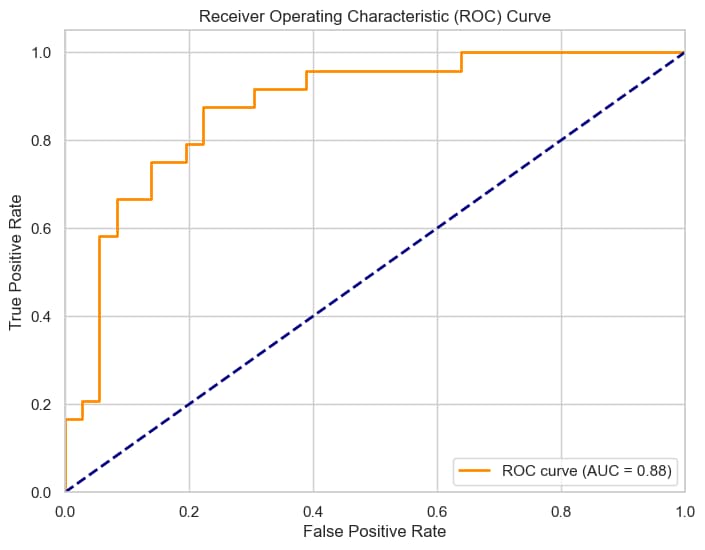
Sistema de Predicción de Enfermedades Cardíacas
Implementación completa de machine learning con procesamiento robusto de datos y gestión de modelos. Características principales: - Pipeline automatizado de datos que maneja valores faltantes y escalado de características - División estratificada de datos con balanceo de clases - Seguimiento de rendimiento del modelo (Precisión, Recall, AUC-ROC) - Despliegue Dockerizado con gestión de dependencias Poetry - Versionado automático de modelos con serialización timestamp - Métricas de evaluación comprehensivas y reproductibilidad

Detalles Técnicos
/src/data/data_loader.py
1 | import pandas as pd
2 |
3 | def load_data(file_path: str) -> pd.DataFrame:
4 | """
5 | Carga los datos desde un archivo CSV.
6 |
7 | Args:
8 | file_path (str): Ruta del archivo CSV.
9 |
10 | Returns:
11 | pd.DataFrame: DataFrame con los datos cargados.
12 | """
13 | return pd.read_csv(file_path)
14 | 
/src/data/data_processor.py
1 | import pandas as pd
2 | import numpy as np
3 |
4 | def process_data(df: pd.DataFrame, columns_to_impute: list, target_column: str = None) -> tuple[pd.DataFrame, pd.Series]:
5 | """
6 | Procesa los datos:
7 | - Imputa los valores faltantes
8 | - Escalar las variables numéricas
9 |
10 | Args:
11 | df (pd.DataFrame): DataFrame con los datos a procesar.
12 | columns_to_impute (list): Lista de columnas a imputar los valores faltantes.
13 | target_column (str, optional): Nombre de la columna objetivo.
14 |
15 | Returns:
16 | pd.DataFrame: DataFrame con los datos procesados.
17 | pd.Series: Serie con la variable objetivo.
18 | """
19 | # Reemplazar valores 0 por NaN en las columnas a imputar
20 | df[columns_to_impute] = df[columns_to_impute].replace(0, np.nan)
21 |
22 | # Extraer la columna objetivo si es proporcionada
23 | target = df[target_column] if target_column else None
24 |
25 | return df, target
26 | 
/src/data/data_splitter.py
1 | from sklearn.model_selection import train_test_split
2 | import pandas as pd
3 |
4 | def split_data(data: pd.DataFrame, target_column: str, test_size=0.2, random_state=42, stratify: bool = True) -> tuple[pd.DataFrame, pd.DataFrame, pd.Series, pd.Series]:
5 | """
6 | Dividir los datos en conjuntos de entrenamiento y prueba
7 |
8 | Args:
9 | data (pd.DataFrame): Dataframe que contiene los datos a dividir
10 | target_column (str): Nombre de la columna objetivo
11 | test_size (float, optional): Proporción de los datos que quedan en el test. Defaults to 0.2.
12 | random_state (int, optional): Semilla para la aleatoriedad. Defaults to 42.
13 |
14 | Returns:
15 | Tupla: Una tupla que contiene los conjuntos de entrenamiento y prueba
16 | - X_train (pd.DataFrame): Conjunto de entrenamiento de las variables independientes
17 | - X_test (pd.DataFrame): Conjunto de prueba de las variables independientes
18 | - y_train (pd.Series): Conjunto de entrenamiento de la variable objetivo
19 | - y_test (pd.Series): Conjunto de prueba de la variable objetivo
20 | """
21 | X = data.drop(columns=target_column, axis=1)
22 | y = data[target_column]
23 |
24 | # Dividir el conjunto de datos en entrenamiento y prueba
25 | if stratify:
26 | X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=test_size, random_state=random_state, stratify=y)
27 | else:
28 | X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=test_size, random_state=random_state)
29 |
30 | return X_train, X_test, y_train, y_test
31 | /src/main.py
1 | import sys
2 | import os
3 | # Agregar la raíz del proyecto al PYTHONPATH
4 | sys.path.append(os.path.abspath(os.path.join(os.path.dirname(__file__), "..")))
5 | from src.data.data_loader import load_data
6 | from src.data.data_processor import process_data
7 | import pandas as pd
8 | from src.data.data_splitter import split_data
9 | from src.model.trainer import train_model
10 | from src.model.evaluator import evaluate_model
11 | from src.model.saver import save_model
12 |
13 | def main():
14 |
15 | # Cargar los datos
16 | heart_disease = load_data(file_path = "data/raw/heart.csv")
17 |
18 | # Preprocesar los datos
19 | processed_data, target = process_data(
20 | df=heart_disease,
21 | columns_to_impute=['trestbps', 'chol', 'thalach', 'oldpeak'], # columnas que pueden tener NaN
22 | target_column='num' # Cambio para coincidir con la columna objetivo del dataset
23 | )
24 |
25 | # Dividir los datos en conjuntos de entrenamiento y prueba
26 | X_train, X_test, y_train, y_test = split_data(processed_data, target_column='num')
27 |
28 | # Convertir 'y' en binario
29 | y_train_binary = (y_train > 0).astype(int)
30 | y_test_binary = (y_test > 0).astype(int)
31 |
32 | # Entrenar el modelo
33 | model = train_model(X_train=X_train, y_train=y_train_binary)
34 |
35 |
36 |
37 | # Evaluar el modelo
38 | accuracy, precision, recall, f1, auc = evaluate_model(model, test_data=X_test, y_test=y_test_binary)
39 |
40 | # Imprimir las métricas
41 | print(f"Accuracy: {accuracy:.2f}")
42 | print(f"Precision: {precision:.2f}")
43 | print(f"Recall: {recall:.2f}")
44 | print(f"F1: {f1:.2f}")
45 | print(f"AUC: {auc:.2f}")
46 |
47 |
48 | # Guardar el modelo
49 | save_model(model, model_path="models/trained_model")
50 |
51 | if __name__ == "__main__":
52 | main()
53 | /src/model/evaluator.py
1 | from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score, roc_auc_score
2 | import xgboost
3 | import pandas as pd
4 |
5 | def evaluate_model(model: xgboost.sklearn.XGBClassifier, test_data: pd.DataFrame, y_test: pd.Series) -> tuple[float, float, float, float, float]:
6 | """
7 | Función para evaluar el modelo
8 |
9 | Args:
10 | model (xgboost.sklearn.XGBClassifier): Modelo a evaluar
11 | test_data (pd.DataFrame): Datos de prueba
12 | y_test (pd.Series): Etiquetas de prueba
13 |
14 | Returns:
15 | tuple[float, float, float, float, float]: Accuracy, Precision, Recall, F1, AUC
16 | """
17 | y_pred = model.predict(test_data)
18 | y_prob = model.predict_proba(test_data)[:, 1]
19 |
20 | accuracy = accuracy_score(y_test, y_pred)
21 | precision = precision_score(y_test, y_pred)
22 | recall = recall_score(y_test, y_pred)
23 | f1 = f1_score(y_test, y_pred)
24 | auc = roc_auc_score(y_test, y_prob)
25 |
26 | return accuracy, precision, recall, f1, auc
27 | 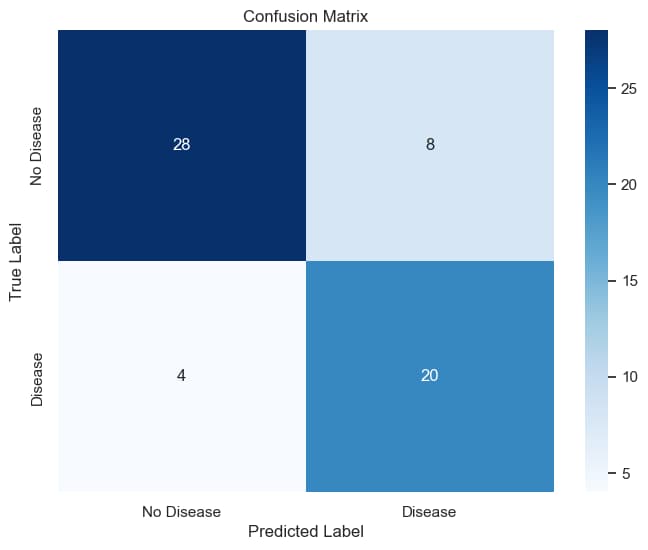
/src/model/saver.py
1 | from datetime import datetime
2 | import joblib
3 | import xgboost
4 |
5 | def save_model(model: xgboost.sklearn.XGBClassifier, model_path: str) -> None:
6 | """
7 | Guarda el modelo en un archivo usando joblib.
8 |
9 | Args:
10 | model (xgboost.sklearn.XGBClassifier): Modelo a guardar.
11 | model_path (str): Ruta donde guardar el modelo (sin extensión).
12 | """
13 | current_date = datetime.now().strftime("%Y-%m-%d")
14 | full_path = f"{model_path}_{current_date}.joblib"
15 | joblib.dump(model, full_path)
16 | print(f"Modelo guardado en {full_path}")
17 | /src/model/trainer.py
1 | import xgboost as xgb
2 | import pandas as pd
3 |
4 | def train_model(X_train: pd.DataFrame, y_train: pd.Series, params: dict = None) -> xgb.sklearn.XGBClassifier:
5 | """
6 | Función para entrenar el modelo
7 |
8 | Args:
9 | X_train (pd.DataFrame): Dataframe con las variables independientes
10 | y_train (pd.Series): Serie con la variable objetivo
11 | params (dict, optional): Diccionario con los parámetros del modelo. Defaults to None.
12 |
13 | Returns:
14 | xgboost.sklearn.XGBClassifier: Modelo entrenado
15 | """
16 | if params is None:
17 | params = {'objective': 'binary:logistic',
18 | 'eval_metric': 'logloss',
19 | 'n_estimators': 300,
20 | 'max_depth': 2,
21 | 'learning_rate': 0.1,
22 | 'random_state': 42}
23 |
24 | model = xgb.XGBClassifier(**params)
25 | model.fit(X_train, y_train)
26 |
27 | return model
28 |